全转录组测序分析
产品简介
全转录组是指特定组织或细胞在特定状态下转录出的所有转录本的总和,包括了mRNA和非编码RNA,非编码RNA包含lncRNA,miRNA,circRNA等。通过构建两种文库(small RNA文库,去rRNA的链特异性文库),或构建三种文库(small RNA文库,去rRNA的链特异性文库,circRNA文库),对四种RNA(miRNA,lncRNA,mRNA,circRNA)的表达及调控关系进行分析,全面构建精细的RNA调控网络,
全转录组测序可实现编码RNA和非编码RNA的分析及联合分析,从而快速全面准确地获得与特定生物学过程(例如发育、疾病等)主要RNA数据信息,将调控机制研究从单一层面延伸到网络立体模式,目标是了解不同类型转录产物之间的行为和机制,分析内容更加全面,可靠性更高,可广泛应用于免疫机制、疾病领域、发育进化研究、致病机理和药物靶标等研究。
技术路线
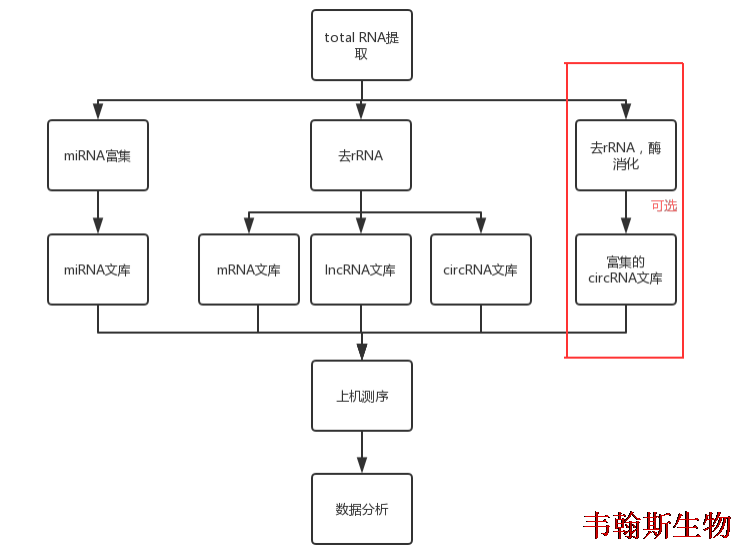
服务内容
|
mRNA分析内容
|
|
基本分析
|
高级分析
|
|
原始数据质控及评估
|
SNP分析(需达到10G数据量)
|
|
比对到参考基因组及结果评估
|
新转录本预测(需达到10G数据量)
|
|
rRNA残留分析
|
可变剪切分析(需达到10G数据量)
|
|
饱和度分析
|
融合基因分析(需达到10G数据量)
|
|
数据分布均一性分析
|
lncRNA分析(需达到10G数据量)
|
|
基因覆盖统计分析
|
差异基因蛋白互作分析
|
|
基因表达定量分析
|
个性化分析
|
|
基因表达分布分析
|
|
|
基因表达差异分析
|
|
|
差异基因聚类分析
|
|
|
GO和KEGG富集分析
|
|
|
lncRNA分析内容
|
|
基本分析
|
高级分析
|
|
原始数据质控及评估
|
与mRNA关联分析(需有mRNA数据)
|
|
lcnRNA表达水平分析
|
lncRNA靶基因预测
|
|
lcnRNA差异表达分析
|
个性化分析
|
|
GO和KEGG富集分析
|
|
|
新lcnRNA预测
|
|
|
miRNA分析内容
|
|
基本分析
|
高级分析
|
|
原始数据质控及评估
|
与mRNA关联分析(需有mRNA数据)
|
|
与miRNA数据库比对分析
|
与lncRNA关联分析(需有lncRNA数据)
|
|
小RNA分类及注释
|
miRNA靶基因预测
|
|
差异表达分析
|
个性化分析
|
|
GO和KEGG富集分析
|
|
|
circRNA分析内容
|
|
基本分析
|
高级分析
|
|
原始数据质控及评估
|
与miRNA关联分析(需有miRNA数据)
|
|
与数据库比对分析
|
个性化分析
|
|
circRNA鉴定与注释
|
|
|
circRNA表达水平分析
|
|
|
鉴定新的circRNA
|
|
|
可变剪切事件识别
|
|
|
circRNA差异表达分析
|
|
|
GO和KEGG富集分析
|
|
|
全转录组分析内容
|
|
其他分析
|
两两关联分析,全关联分析,个性化分析
|
案例展示
Identification of Novel Long Non-coding and Circular RNAs in Human Papillomavirus-Mediated Cervical Cancer
鉴定宫颈癌lncRNA和环状RNA等biomarker及ceRNA分析
研究人员选取3个病人(HPV16)的宫颈鳞癌组织和癌旁组织,采用两种建库方式,通过全转录组测序来研究宫颈癌lncRNA和环状RNA致病分子调控机制。
全转录组测序结果经过分析,共鉴定到差异表达的19个lncRNA,99个环状RNA,304个mRNA。非编码RNA中鉴定到3个新lncRNA和44个新环状RNA,并对这些差异RNA进行功能富集分析。共表达分析和功能预测中,发现19个差异表达lncRNA在致癌和癌症发展中具有重要作用;另外还研究了ceRNA网络分析表达和非表达RNA的相互作用,及每一个miRNA靶向作用的lncRNA和Mrna竞争关系。研究结果表明,非编码RNA将有可能作为宫颈鳞癌治疗和诊断的biomarker。CeRNA网络研究在未来宫颈鳞癌研究中将对编码RNA和非编码RNA的关系及重要作用研究的将是重要的分析工具。
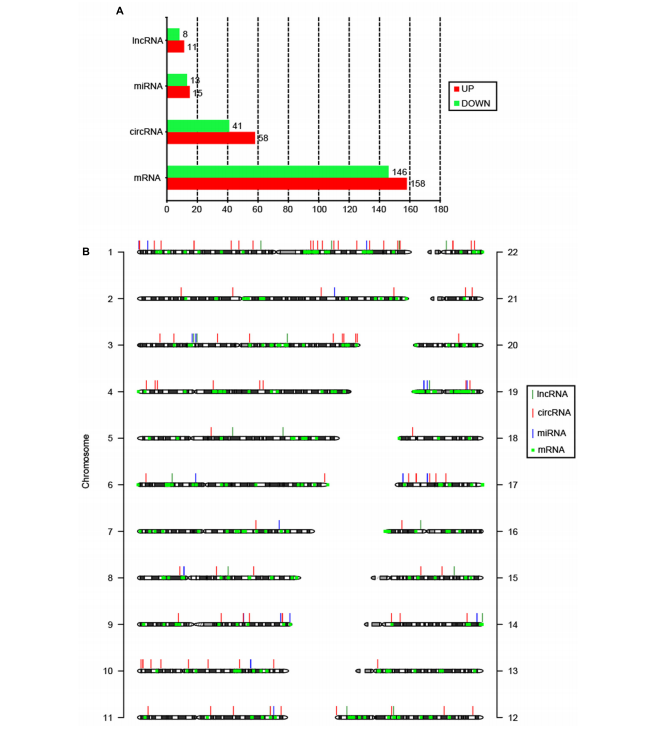
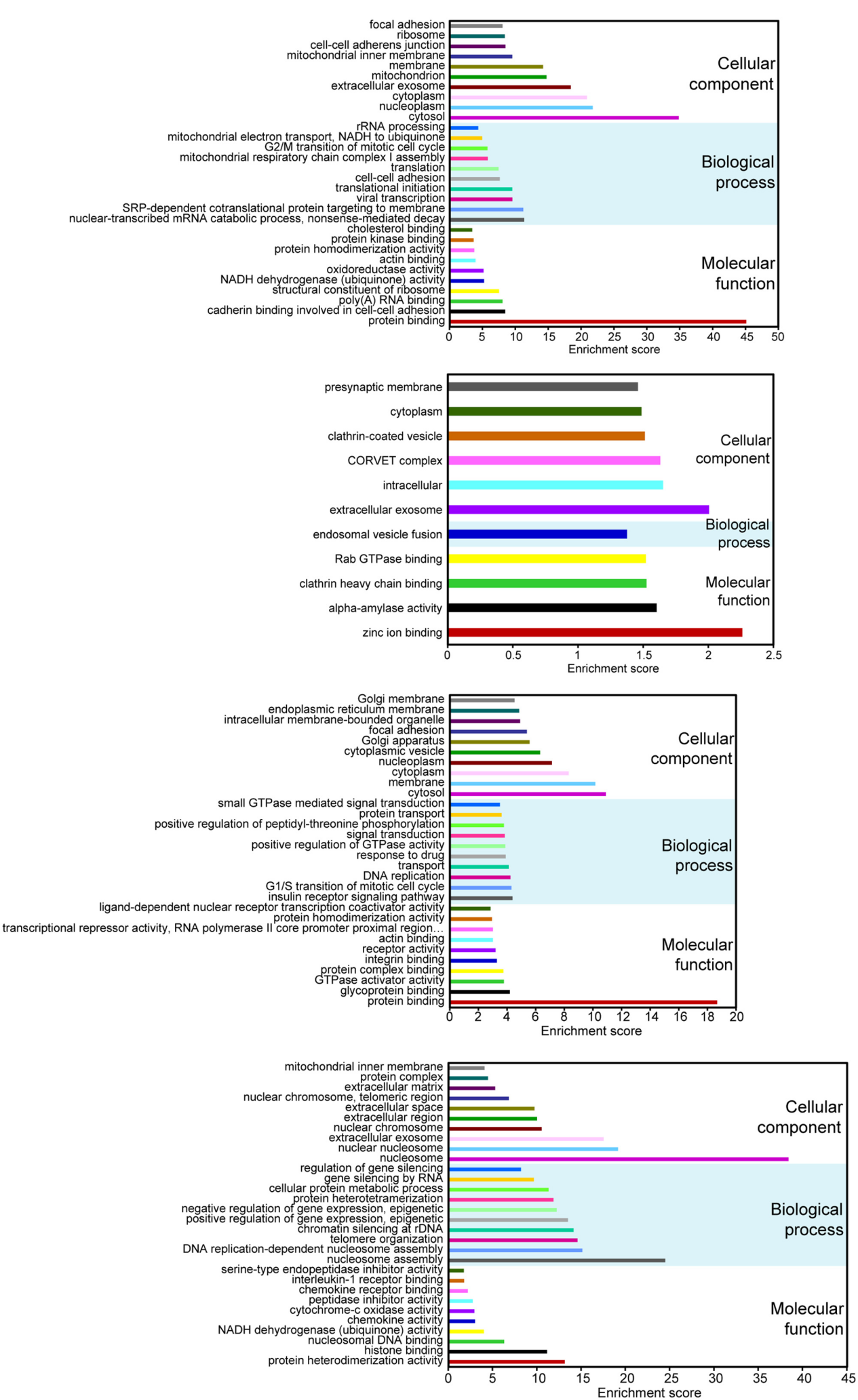
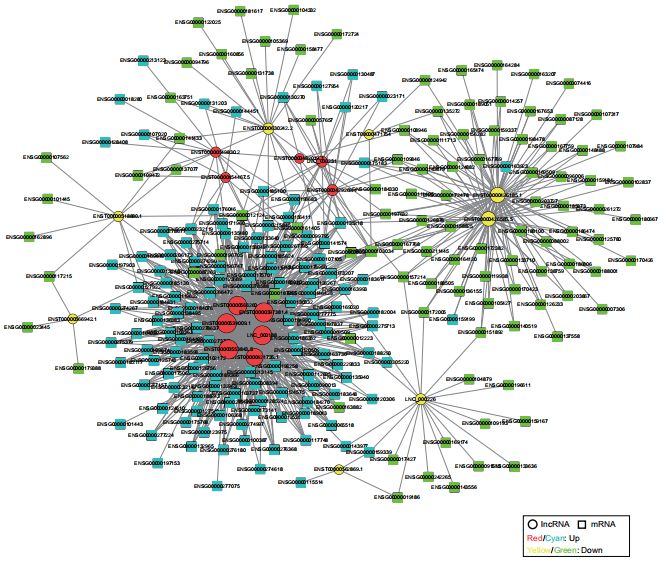
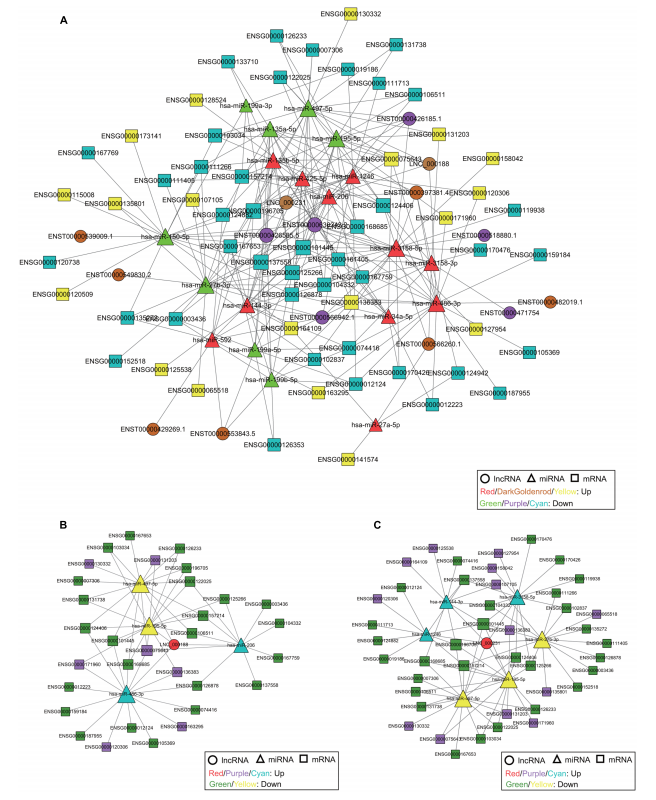
Wang H, Zhao Y, Chen M and Cui J (2017) Identification of Novel Long Non-coding and Circular RNAs in Human Papillomavirus-Mediated Cervical Cancer.Front. Microbiol. 8:1720.

